The genus trace: a function that shows values of genus (vertical axis) for subchains spanned between the first residue, and all other residues (shown on horizontal axis). The number of the latter residue and the genus of a given subchain are shown interactively.
Total Genus |
41
|
sequence length |
153
|
structure length |
150
|
Chain Sequence |
TTRSAEHTTFVIERRLTAPVARVFRAWSTPESKRQWFACHVPLEYALDFRPGGTERNYTADTDGLLHAYDARYIDIVPDTRIIYAYEMKLGQTRISASLVTVAFDVEPSGTRMVFTEQVVFLDGYGDNGARLQGTEIGLDNLELFLVRET
|
The genus matrix. At position (x,y) a genus value for a subchain spanned between x’th and y’th residue is shown. Values of the genus are represented by color, according to the scale given on the right.
After clicking on a point (x,y) in the genus matrix above, a subchain from x to y is shown in color.
| publication title |
Crystal Structure of the CERT START domain (mutant V151E) from Rhizobium etli at the resolution 1.8A, Northeast Structural Genomics Consortium Target ReR239.
rcsb |
| molecule tags |
Structure genomics, unknown function
|
| source organism |
Rhizobium etli cfn 42
|
| molecule keywords |
Hypothetical conserved protein
|
| total genus |
41
|
| structure length |
150
|
| sequence length |
153
|
| chains with identical sequence |
B
|
| ec nomenclature | |
| pdb deposition date | 2010-12-06 |
| chain | Pfam Accession Code | Pfam Family Identifier | Pfam Description |
|---|---|---|---|
| A | PF08327 | AHSA1 | Activator of Hsp90 ATPase homolog 1-like protein |
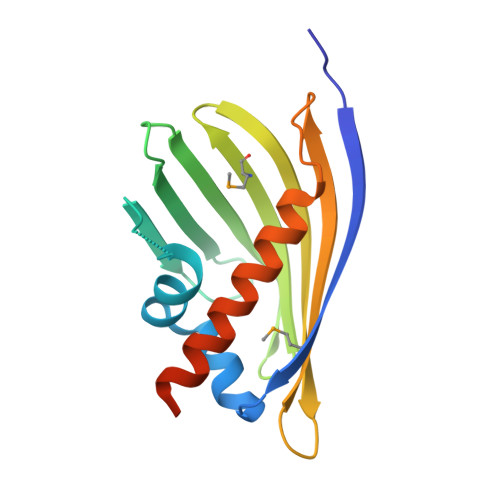
 Image from the rcsb pdb (www.rcsb.org)
Image from the rcsb pdb (www.rcsb.org)
cath code
| Class | Architecture | Topology | Homology | Domain |
|---|---|---|---|---|---|
| Alpha Beta | 2-Layer Sandwich | Alpha-D-Glucose-1,6-Bisphosphate; Chain A, domain 4 | Alpha-D-Glucose-1,6-Bisphosphate; Chain A, domain 4 |
#chains in the Genus database with same CATH superfamily 2REZ A; 4QIP A; 3KDH A; 2LF2 A; 1EM2 A; 3H3Q A; 4LG5 A; 3JRQ B; 2E3Q A; 3KLX A; 2L65 A; 2RES A; 3Q6A A; 4R7K A; 5I9J A; 4A85 A; 4DSC A; 4OIC A; 3H3R A; 2E3S A; 4XRT A; 3TVQ A; 1LLT A; 2FFS A; 4IHR A; 2PSO A; 2E3N A; 2LGH A; 3IJT A; 2N4B A; 1Z94 A; 2D4R A; 3PUT A; 4M9B A; 4JHG A; 4MNS A; 2E3O A; 4A8U A; 3NJ0 A; 3P51 A; 4A83 A; 1FSK A; 2LE1 A; 4M9W A; 5BRL A; 4JHI A; 3KDJ A; 3NI8 A; 3W9K A; 4Z3L A; 4N3E A; 2WQL A; 3NMH A; 4LGA A; 4BKC A; 3NMP A; 4A80 A; 3WG8 A; 1LN1 A; 2MOU A; 2LEQ A; 2KTE A; 3NEF A; 2N4A A; 2LCG A; 1VJH A; 1ZXF A; 2KCZ A; 3RD6 A; 3UQH A; 4C9C A; 3ELI A; 1LN3 A; 3H3S A; 4Y31 A; 5C9Y A; 5E46 A; 2LPX A; 3KB0 A; 4PSB A; 2R55 A; 4REJ A; 4C94 A; 3H3T A; 2RER A; 3TL1 A; 2Q3Q A; 2IL5 A; 1UW5 A; 1LN2 A; 1XFS A; 3OJI A; 3PU2 A; 3RT0 C; 3NMN A; 5E4B A; 3K90 A; 4IH0 A; 2K7H A; 3TFZ A; 4A8G A; 1FM4 A; 3UID A; 3NJO A; 2VNE A; 3NMV A; 3KDI A; 3P0L A; 1TW0 A; 2LUZ A; 3P9V A; 4REI A; 3KAZ A; 3NJ1 A; 4RYV A; 4WVO A; 3CNW A; 2BK0 A; 2K5G A; 3JRS A; 3KL1 A; 4MA6 A; 3UJL A; 5I8F A; 2KF2 A; 2LDK A; 3F08 A; 3IE5 A; 4JDA A; 4GY9 A; 3C0V A; 4LA7 A; 1ICX A; 2M89 A; 2I9Y A; 1XN6 A; 4A81 A; 2EJX A; 1QMR A; 2Z9Z A; 3NEG A; 3NS2 A; 4BTZ A; 2LAK A; 3NMT A; 5JNN A; 2LIO A; 1H2O A; 2A1L A; 3R6P A; 3FO5 A; 4N0G C; 5AMW A; 1IFV A; 4B9R A; 1B6F A; 4C9I A; 4JDL A; 2E3R A; 1T17 A; 3ZVU A; 4XRW A; 3OQU A; 4FPW A; 3GGN A; 4BK7 A; 3Q63 A; 3TVR A; 2GKD A; 4JHH A; 3KB3 A; 4A88 A; 4BK6 A; 2FLH A; 4IGW A; 4Q0K A; 2NS9 A; 2KEW A; 3Q64 A; 3QN1 A; 4A8V A; 3RT2 A; 2QPV A; 1XDF A; 2NN5 A; 2VQ5 A; 4BKD A; 3K3K A; 4IH2 A; 3OH8 A; 2PCS A; 3W9R A; 1E09 A; 4A84 A; 4DS8 A; 2E3P A; 5E4M A; 2Z9Y A; 2E3M A; 2L8O A; 1BTV A; 3KAY A; 4A87 A; 1XUV A; 3QSZ A; 1T27 A; 1BV1 A; 1JSS A; 4DSB A; 1XN5 A; 4IGX A; 1TXC A; 3OTL A; 4IGV A; 3E85 A; 2M47 A; 1X53 A; 1KCM A; 4REH A; 2L9P A; 3NR4 A; 4MAP A; 5E4D A; 5JO2 A; 4A86 A; 2QIM A; 4IGY A; 4LGB A; 5JO1 A; #chains in the Genus database with same CATH topology 2REZ A; 4QIP A; 3KDH A; 2LF2 A; 1EM2 A; 3H3Q A; 4LG5 A; 3JRQ B; 2E3Q A; 3KLX A; 4E6F A; 2L65 A; 2RES A; 3Q6A A; 4R7K A; 3NQN A; 5I9J A; 4QCK A; 4A85 A; 4DSC A; 3GL2 A; 4OIC A; 3H3R A; 2E3S A; 4XRT A; 3TVQ A; 1LLT A; 2FFS A; 4IHR A; 2PSO A; 2E3N A; 2LGH A; 3IJT A; 2N4B A; 3F73 A; 1Z94 A; 2D4R A; 3GKE A; 3PUT A; 4JHG A; 4M9B A; 2E3O A; 4MNS A; 4A8U A; 3NJ0 A; 3P51 A; 4A83 A; 1FSK A; 3GTE A; 2LE1 A; 4M9W A; 5BRL A; 4JHI A; 3KDJ A; 3NI8 A; 3W9K A; 4Z3L A; 4N3E A; 2WQL A; 3NMH A; 4LGA A; 4NCB A; 4BKC A; 3NMP A; 4A80 A; 3WG8 A; 1LN1 A; 2MOU A; 2LEQ A; 2KTE A; 3NEF A; 2N4A A; 2LCG A; 1VJH A; 1ZXF A; 2KCZ A; 3RD6 A; 3UQH A; 4C9C A; 3ELI A; 1LN3 A; 3H3S A; 4Y31 A; 5C9Y A; 5E46 A; 2LPX A; 3KB0 A; 4PSB A; 2R55 A; 4REJ A; 4C94 A; 3GOB A; 3H3T A; 4NCA A; 2RER A; 3TL1 A; 2Q3Q A; 2IL5 A; 1UW5 A; 1LN2 A; 1XFS A; 3OJI A; 3PU2 A; 3RT0 C; 3GL0 A; 3NMN A; 5E4B A; 3K90 A; 4IH0 A; 2K7H A; 3TFZ A; 4A8G A; 1FM4 A; 3UID A; 3NJO A; 2VNE A; 3NMV A; 3KDI A; 3P0L A; 1TW0 A; 2LUZ A; 3P9V A; 4REI A; 3KAZ A; 3NJ1 A; 4RYV A; 4WVO A; 3CNW A; 2BK0 A; 2K5G A; 3JRS A; 3KL1 A; 4MA6 A; 3UJL A; 5I8F A; 2KF2 A; 2LDK A; 3F08 A; 3IE5 A; 4JDA A; 4JHY A; 4GY9 A; 3C0V A; 4LA7 A; 1ICX A; 4N76 A; 2M89 A; 2I9Y A; 3HM9 A; 1XN6 A; 4N47 A; 2EJX A; 4A81 A; 1QMR A; 2LAF A; 2Z9Z A; 3NEG A; 3NS2 A; 4QDF B; 4BTZ A; 2LAK A; 3NMT A; 5JNN A; 2LIO A; 1H2O A; 2A1L A; 3GB4 A; 3FO5 A; 3R6P A; 4N0G C; 1IFV A; 4QDF A; 5AMW A; 3HJF A; 4B9R A; 1B6F A; 3TGO C; 3HXM A; 2YH6 A; 4C9I A; 4JDL A; 2E3R A; 1T17 A; 3GTS A; 4XRW A; 3ZVU A; 3OQU A; 4FPW A; 3GGN A; 4BK7 A; 3Q63 A; 3TVR A; 2GKD A; 4JHH A; 3KB3 A; 4A88 A; 4BK6 A; 3HVR A; 2FLH A; 4IGW A; 4Q0K A; 2NS9 A; 2KEW A; 3Q64 A; 3HO1 A; 3QN1 A; 4A8V A; 3RT2 A; 2QPV A; 1XDF A; 2NN5 A; 2VQ5 A; 4BKD A; 4QDC A; 2ZYL A; 3K3K A; 4IH2 A; 3OH8 A; 2PCS A; 3W9R A; 1E09 A; 4A84 A; 4DS8 A; 2E3P A; 5E4M A; 2Z9Y A; 2E3M A; 4QDD A; 2L8O A; 1BTV A; 3KAY A; 4A87 A; 1XUV A; 3QSZ A; 1T27 A; 1BV1 A; 1JSS A; 4DSB A; 1XN5 A; 4KPY A; 4IGX A; 3DLB A; 3HK2 A; 1TXC A; 3OTL A; 4IGV A; 3E85 A; 2M47 A; 1X53 A; 1KCM A; 4REH A; 5EKQ C; 2L9P A; 3NR4 A; 4MAP A; 5E4D A; 5JO2 A; 4A86 A; 3DLH A; 2QIM A; 4IGY A; 4LGB A; 5JO1 A; 4N41 A; #chains in the Genus database with same CATH homology 2REZ A; 4QIP A; 3KDH A; 2LF2 A; 1EM2 A; 3H3Q A; 4LG5 A; 3JRQ B; 2E3Q A; 3KLX A; 4E6F A; 2L65 A; 2RES A; 3Q6A A; 4R7K A; 3NQN A; 5I9J A; 4QCK A; 4A85 A; 4DSC A; 3GL2 A; 4OIC A; 3H3R A; 2E3S A; 4XRT A; 3TVQ A; 1LLT A; 2FFS A; 4IHR A; 2PSO A; 2E3N A; 2LGH A; 3IJT A; 2N4B A; 3F73 A; 1Z94 A; 2D4R A; 3GKE A; 3PUT A; 4JHG A; 4M9B A; 2E3O A; 4MNS A; 4A8U A; 3NJ0 A; 3P51 A; 4A83 A; 1FSK A; 3GTE A; 2LE1 A; 4M9W A; 5BRL A; 4JHI A; 3KDJ A; 3NI8 A; 3W9K A; 4Z3L A; 4N3E A; 2WQL A; 3NMH A; 4LGA A; 4NCB A; 4BKC A; 3NMP A; 4A80 A; 3WG8 A; 1LN1 A; 2MOU A; 2LEQ A; 2KTE A; 3NEF A; 2N4A A; 2LCG A; 1VJH A; 1ZXF A; 2KCZ A; 3RD6 A; 3UQH A; 4C9C A; 3ELI A; 1LN3 A; 3H3S A; 4Y31 A; 5C9Y A; 5E46 A; 2LPX A; 3KB0 A; 4PSB A; 2R55 A; 4REJ A; 4C94 A; 3GOB A; 3H3T A; 4NCA A; 2RER A; 3TL1 A; 2Q3Q A; 2IL5 A; 1UW5 A; 1LN2 A; 1XFS A; 3OJI A; 3PU2 A; 3RT0 C; 3GL0 A; 3NMN A; 5E4B A; 3K90 A; 4IH0 A; 2K7H A; 3TFZ A; 4A8G A; 1FM4 A; 3UID A; 3NJO A; 2VNE A; 3NMV A; 3KDI A; 3P0L A; 1TW0 A; 2LUZ A; 3P9V A; 4REI A; 3KAZ A; 3NJ1 A; 4RYV A; 4WVO A; 3CNW A; 2BK0 A; 2K5G A; 3JRS A; 3KL1 A; 4MA6 A; 3UJL A; 5I8F A; 2KF2 A; 2LDK A; 3F08 A; 3IE5 A; 4JDA A; 4JHY A; 4GY9 A; 3C0V A; 4LA7 A; 1ICX A; 4N76 A; 2M89 A; 2I9Y A; 3HM9 A; 1XN6 A; 4N47 A; 2EJX A; 4A81 A; 1QMR A; 2LAF A; 2Z9Z A; 3NEG A; 3NS2 A; 4QDF B; 4BTZ A; 2LAK A; 3NMT A; 5JNN A; 2LIO A; 1H2O A; 2A1L A; 3GB4 A; 3FO5 A; 3R6P A; 4N0G C; 1IFV A; 4QDF A; 5AMW A; 3HJF A; 4B9R A; 1B6F A; 3TGO C; 3HXM A; 2YH6 A; 4C9I A; 4JDL A; 2E3R A; 1T17 A; 3GTS A; 4XRW A; 3ZVU A; 3OQU A; 4FPW A; 3GGN A; 4BK7 A; 3Q63 A; 3TVR A; 2GKD A; 4JHH A; 3KB3 A; 4A88 A; 4BK6 A; 3HVR A; 2FLH A; 4IGW A; 4Q0K A; 2NS9 A; 2KEW A; 3Q64 A; 3HO1 A; 3QN1 A; 4A8V A; 3RT2 A; 2QPV A; 1XDF A; 2NN5 A; 2VQ5 A; 4BKD A; 4QDC A; 2ZYL A; 3K3K A; 4IH2 A; 3OH8 A; 2PCS A; 3W9R A; 1E09 A; 4A84 A; 4DS8 A; 2E3P A; 5E4M A; 2Z9Y A; 2E3M A; 4QDD A; 2L8O A; 1BTV A; 3KAY A; 4A87 A; 1XUV A; 3QSZ A; 1T27 A; 1BV1 A; 1JSS A; 4DSB A; 1XN5 A; 4KPY A; 4IGX A; 3DLB A; 3HK2 A; 1TXC A; 3OTL A; 4IGV A; 3E85 A; 2M47 A; 1X53 A; 1KCM A; 4REH A; 5EKQ C; 2L9P A; 3NR4 A; 4MAP A; 5E4D A; 5JO2 A; 4A86 A; 3DLH A; 2QIM A; 4IGY A; 4LGB A; 5JO1 A; 4N41 A;
#similar chains in the Genus database (?% sequence similarity) ...loading similar chains, please wait... #similar chains, but unknotted ...loading similar chains, please wait... #similar chains in the pdb database (?% sequence similarity) ...loading similar chains, please wait...